Nature Methods | 昌平实验室高毅勤等团队整合多种实验信息利用GRASP辅助蛋白质复合物结构预测
iNature
2025-09-16 19:11
文章摘要
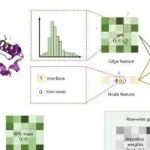
背景:蛋白质-蛋白质相互作用在生物过程中至关重要,但现有计算方法如AlphaFold3等在预测精度上仍存在局限,而各种实验方法提供的零散信息需要有效整合工具。研究目的:昌平实验室团队开发了GRASP模型,旨在高效整合交联质谱、共价标记、化学位移扰动等多种实验数据,提升蛋白质复合物结构预测的准确性和灵活性。结论:GRASP在模拟和真实实验约束下均优于现有工具,尤其在抗原抗体复合物预测中表现突出,甚至超越AlphaFold3,并展示了在近细胞条件下建模蛋白质相互作用组的潜力,为复杂生物系统研究提供了可靠工具。
本站注明稿件来源为其他媒体的文/图等稿件均为转载稿,本站转载出于非商业性的教育和科研之目的,并不意味着赞同其观点或证实其内容的真实性。如转载稿涉及版权等问题,请作者速来电或来函联系。
iNature
最新文章

中山大学孙颖/马骏等合作最新Nature
9小时前






