Nat Methods|GRASP:整合多样实验信息助力蛋白质复合物结构预测
智药邦
2025-09-24 08:00
文章摘要
背景:蛋白质-蛋白质相互作用在生物过程中至关重要,但现有预测方法如AlphaFold-Multimer在复合物预测中精度有限,实验方法提供的信息零散不完整。研究目的:开发GRASP通用框架,高效整合残基对约束和界面约束等多种实验数据提升预测精度。结论:GRASP在模拟和真实数据中均优于现有方法,特别在抗原-抗体预测中超越AlphaFold3,支持多源约束联合建模,并展示了在线粒体原位互作网络解析的应用潜力。
本站注明稿件来源为其他媒体的文/图等稿件均为转载稿,本站转载出于非商业性的教育和科研之目的,并不意味着赞同其观点或证实其内容的真实性。如转载稿涉及版权等问题,请作者速来电或来函联系。
推荐文献
Metabolism of Epigenetic Ribonucleosides Leads to Nucleolar Stress and Cytotoxicity.
DOI: 10.1021/acschembio.5c00656
Pub Date : 2026-03-06
Large-scale library of 3D ant images captured with synchrotron X-ray microtomography.
DOI: 10.1038/s41592-026-03004-1
Pub Date : 2026-03-05
OSCAR: A Modular Open-Source Robotic Platform for Biological Laboratories.
DOI: 10.1021/acssynbio.5c00733
Pub Date : 2026-03-09
智药邦

关于账号进行迁移的说明.
2025-10-20

专家点评Cell | 卢培龙团队及其合作者从头设计新型电压门控阴离子通道.
2025-10-20
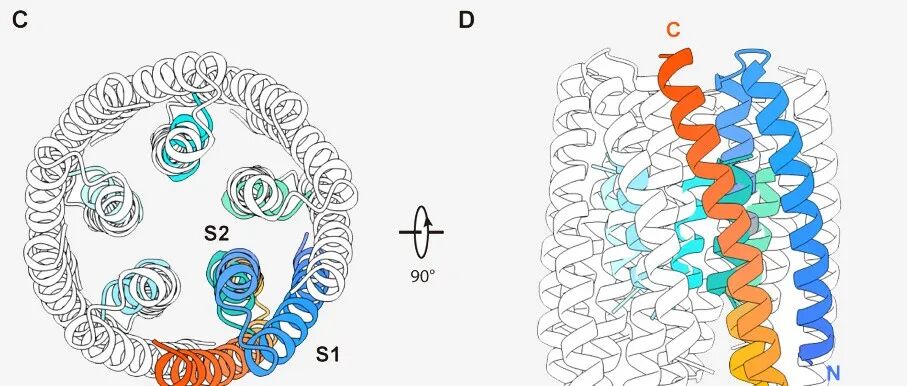
Cell|西湖大学卢培龙/黄晶等:从头设计新型电压门控阴离子通道.
2025-10-20

Sci Adv丨陈洛南课题组为计算生物学中的干预性因果推断提供新思路.
2025-10-20
Nature|麻省理工学院李巨团队:一种用于多组分电催化剂筛选的多模态机器人平台.
2025-10-19
最新文章

关于账号进行迁移的说明
2025-10-20

Sci Adv丨陈洛南课题组为计算生物学中的干预性因果推断提供新思路
2025-10-20

Cell|西湖大学卢培龙/黄晶等:从头设计新型电压门控阴离子通道
2025-10-20

专家点评Cell | 卢培龙团队及其合作者从头设计新型电压门控阴离子通道
2025-10-20




