Nat Commun丨香港大学黄渊华等团队研究发现CLADES可揭示克隆细胞命运和分化动力学
iNature
2025-09-05 00:05
文章摘要
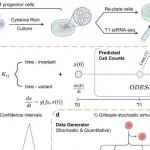
背景:基于谱系追踪的单细胞技术(LT-scSeq)如LARRY条形码系统已用于解析克隆层面的分化轨迹,但克隆特异性动力学的异质性仍缺乏定量研究。研究目的:香港大学黄渊华等团队开发了CLADES框架,结合NeuralODE和随机模拟算法,旨在从LT-scSeq数据中准确估算克隆和群体特异性动力学,重建祖细胞谱系树,并分析克隆水平行为。结论:CLADES能有效量化细胞群体的克隆特异性分化动力学,揭示造血干细胞和祖细胞在不同转录状态下的特异性行为,为发育生物学研究提供了可扩展的定量分析方法。
本站注明稿件来源为其他媒体的文/图等稿件均为转载稿,本站转载出于非商业性的教育和科研之目的,并不意味着赞同其观点或证实其内容的真实性。如转载稿涉及版权等问题,请作者速来电或来函联系。
推荐文献
Global burden of non-communicable diseases attributable to behavioral factors.
DOI: 10.1016/j.scib.2025.08.037
Pub Date : 2025-08-20
Out of the cave: Rewilding deep time at the Venice Biennale
DOI: 10.1016/j.isci.2025.113392
Pub Date : 2025-09-02
Many plants naturalized as aliens abroad have also become more common within their native regions
DOI: 10.1038/s41467-025-63293-6
Pub Date : 2025-09-05
iNature
最新文章
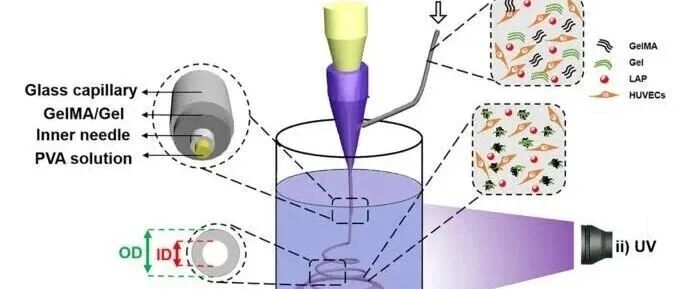
GelMA应用:微流控
10小时前