文献分享| Nature Methods: 使用 LigandMPNN 的原子环境条件蛋白序列设计
YaolabHNU
2025-04-10 18:12
文章摘要
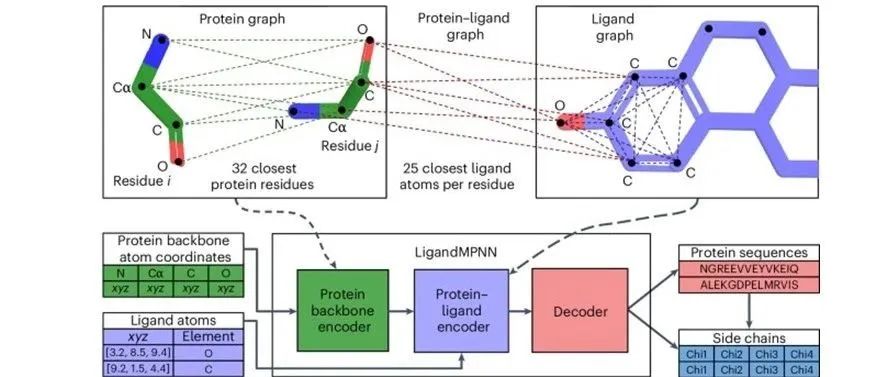
本文介绍了David Baker团队开发的一种基于深度学习的蛋白质序列设计方法LigandMPNN。该方法针对小分子、核苷酸和金属离子的蛋白质序列设计,显著优于传统的Rosetta和ProteinMPNN方法。LigandMPNN不仅能生成序列,还能生成侧链构象,已成功设计出100多种经实验验证的小分子和DNA结合蛋白。文章详细阐述了LigandMPNN的模型架构、计算机评估和实验验证结果,并展望了其在蛋白质设计领域的广泛应用前景。
本站注明稿件来源为其他媒体的文/图等稿件均为转载稿,本站转载出于非商业性的教育和科研之目的,并不意味着赞同其观点或证实其内容的真实性。如转载稿涉及版权等问题,请作者速来电或来函联系。
推荐文献
Take a dive into seascape genomics.
DOI: 10.1038/s41592-026-03002-3
Pub Date : 2026-01-26
Enzymatic Synthesis of 2-Amino-adenosine Triphosphate as a Noncanonical Nucleotide Precursor of Z-Modified RNA.
DOI: 10.1021/acssynbio.5c00890
Pub Date : 2026-02-06
Protocol for Discovery and Characterization of Miniature Cas12 Systems.
DOI: 10.1021/acschembio.6c00016
Pub Date : 2026-02-06









