复旦大学王满宁团队提出ProteinF3S模型,通过融合蛋白序列、结构和表面信息进行酶分类预测
智药邦
2025-03-17 18:03
文章摘要
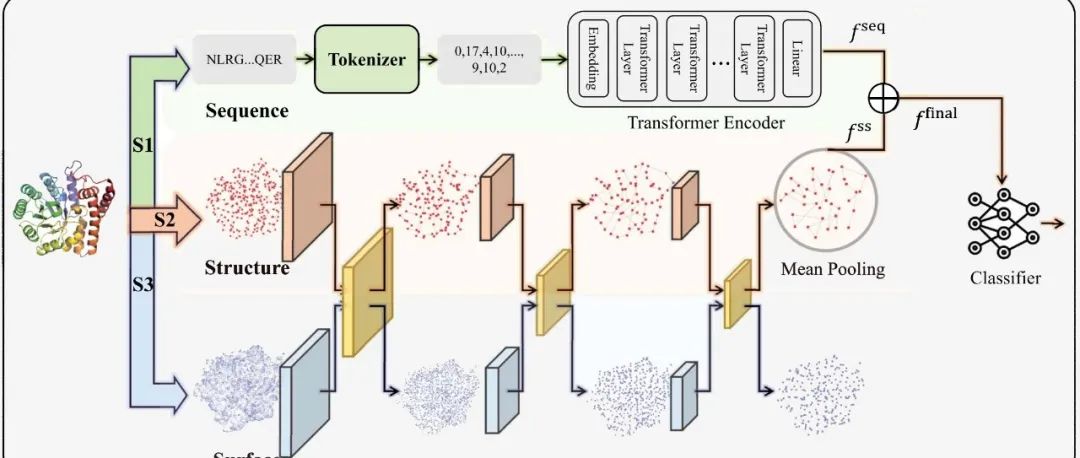
复旦大学王满宁团队在Briefings in Bioinformatics上发表文章,提出了一种名为ProteinF3S的新框架,该框架通过融合蛋白质的序列、结构和表面信息来增强酶功能预测的准确性。研究背景在于蛋白质表面信息在以往的研究中较少被采用,但其在原子水平上提供了更清晰的局部特性建模。研究目的是通过多尺度双向融合策略,将序列特征与结构和表面信息进行串联,以实现更有效的融合。实验结果表明,ProteinF3S在酶分类预测任务上的性能超越了现有方法。结论指出,融合不同形式的蛋白质信息确实可以提高性能,并且融合策略在提升模型性能中起着关键作用。未来,ProteinF3S有望扩展到更多的蛋白质任务中。
本站注明稿件来源为其他媒体的文/图等稿件均为转载稿,本站转载出于非商业性的教育和科研之目的,并不意味着赞同其观点或证实其内容的真实性。如转载稿涉及版权等问题,请作者速来电或来函联系。
推荐文献
Benchmarking community drug response prediction models: datasets, models, tools, and metrics for cross-dataset generalization analysis.
DOI: 10.1093/bib/bbaf667
Pub Date : 2026-01-07
Multilevel Regulation in RNA-Protein Hybrid Incoherent Feed-Forward Loop Circuits for Tunable Pulse Dynamics in <i>Escherichia coli</i>.
DOI: 10.1021/acssynbio.5c00111
Pub Date : 2026-02-03
Three-Component Glycosylation of Transient Hemiacetals Toward Tunable Aryl-Bisacetal Substrates.
DOI: 10.1021/acschembio.5c01026
Pub Date : 2026-02-04
智药邦

关于账号进行迁移的说明.
2025-10-20

专家点评Cell | 卢培龙团队及其合作者从头设计新型电压门控阴离子通道.
2025-10-20
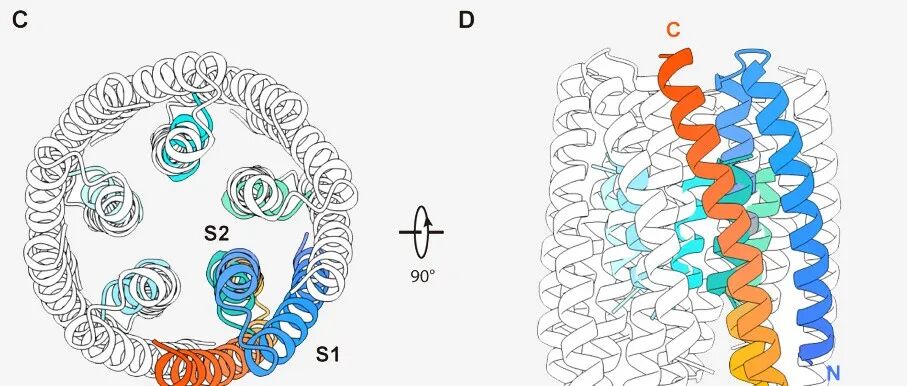
Cell|西湖大学卢培龙/黄晶等:从头设计新型电压门控阴离子通道.
2025-10-20

Sci Adv丨陈洛南课题组为计算生物学中的干预性因果推断提供新思路.
2025-10-20
Nature|麻省理工学院李巨团队:一种用于多组分电催化剂筛选的多模态机器人平台.
2025-10-19
最新文章

关于账号进行迁移的说明
2025-10-20

Sci Adv丨陈洛南课题组为计算生物学中的干预性因果推断提供新思路
2025-10-20

Cell|西湖大学卢培龙/黄晶等:从头设计新型电压门控阴离子通道
2025-10-20

专家点评Cell | 卢培龙团队及其合作者从头设计新型电压门控阴离子通道
2025-10-20




