研究论文: 蛋白质解折叠的拉伸分子动力学模拟效率优化
中国高分子
2024-09-27 12:00
文章摘要
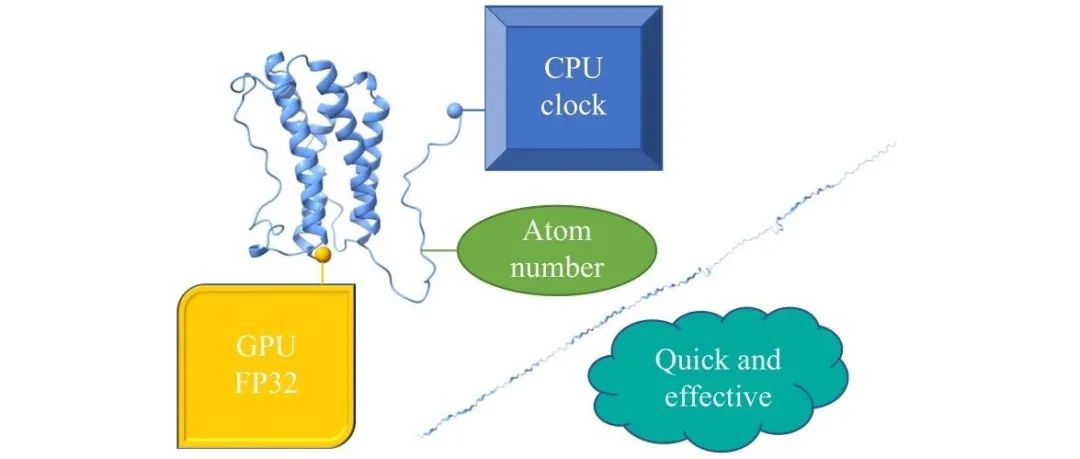
南京大学郑鹏团队研究了使用GROMACS进行拉伸分子动力学模拟(SMD)的最佳硬件配置。研究表明,八核中央处理器(CPU)与一块图形处理器(GPU)的配置能提供最高效的性能。具有更高频率的CPU和更强FP32计算能力的GPU可以显著提升模拟效果。此外,模拟体系原子数与模拟效率呈负相关,体系原子数小于1.2×10^7时这种相关性较为明显。该研究为未来利用分子动力学研究更复杂的高分子力学行为提供了重要信息。
本站注明稿件来源为其他媒体的文/图等稿件均为转载稿,本站转载出于非商业性的教育和科研之目的,并不意味着赞同其观点或证实其内容的真实性。如转载稿涉及版权等问题,请作者速来电或来函联系。
中国高分子

中科院宁波材料所陈涛、乐晓霞团队 | 智能聚合物凝胶信息存储材料在防伪领域的研究进展.
2025-12-31

功能高分子学报 | 2025年度封面文章汇总.
2025-12-31

聚合新篇,链动未来 | 高分子三刊编辑部祝您新年快乐,科研花开!.
2025-12-30

高分子通报2025年度最美封面评选.
2025-12-30

快来投票!高分子学报2025年度最美封面评选等你来.
2025-12-30
最新文章

《AM》西南交通大学:面向下一代个性化医疗的离子水凝胶传感器
2026-01-23

沸腾!金属有机框架(MOFs)基水凝胶的最新研究进展,破百年难题!
2026-01-23

《AFM》燕山大学张强:筋膜仿生核壳纳米纤维水凝胶,具有免疫调节与疤痕抑制双重功效!
2026-01-22


